9、
さらに隣り合う細胞を区別してカウントされるように [Watershed] の処理を行います。 この処理を実行すると隣接した細胞を分割することができます。
さらに隣り合う細胞を区別してカウントされるように [Watershed] の処理を行います。 この処理を実行すると隣接した細胞を分割することができます。
10、
Analyze→Analyze Particlesを選択します。Sizeを5 μm2-infinity、Circularity (真円率 0.00-1.00) 、Show: Outlines、Summarizeにチェック☑を入れOKをクリックします。
Analyze→Analyze Particlesを選択します。Sizeを5 μm2-infinity、Circularity (真円率 0.00-1.00) 、Show: Outlines、Summarizeにチェック☑を入れOKをクリックします。
※真円率とは・・・真円のときが1.0で1.0より小さな値をとるほど円形から遠ざかることを意味する数値です。
11、
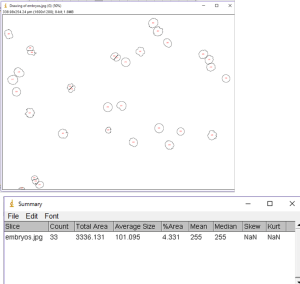
どの細胞を数えたかを示した画像、および細胞数の計測結果を示すSummaryが出力されます。
どの細胞を数えたかを示した画像、および細胞数の計測結果を示すSummaryが出力されます。
【マクロ言語を用いて二値化を施す】
ここでは ≪パターン①≫ Otsu法による自動二値化と ≪パターン②≫ 固定値を閾値とした二値化のスクリプトを紹介します。また画像には対象物が黒、背景が白のものと対象物が黒で背景が白のものがあります。今回の記事では、どちらの画像に対してもProcess→Binary→Options にある “Black Background” (背景を黒に) のチェックを外した状態で説明します (*次回の記事で詳しく説明します)。
≪パターン①≫ Otsu法による自動二値化
◆対象物が黒、背景が白
◆対象物が黒、背景が白
※上記のスクリプトでは細胞の大きさの範囲やスケールについてあらかじめ既知であるとして記述しています。スケールの設定をしないときは細胞の面積の指定はピクセル単位 (pixels) になるためrun ("set Scale..."以降のスクリプトは以下のようになります。細胞以外の黒い小さな点を測定すると20 pixels程度になるため、粒子の面積の範囲を100-infinity (単位; pixels) と設定します。結果は34個になるのでスケールを設定した場合と同じになります。











 エルピクセル編集部
エルピクセル編集部





