今回は人工知能、ディープラーニングで細胞の種類を判別してみたいと思っています。判別するのは私が普段実験で使っている上皮細胞のEpH4という細胞と、遺伝子発現用のHEK293という細胞です。
見ると結構見た目が違います。この特徴を学習させます。
機械学習のフレームワークにはいろいろなものがありますが、今回はchainerを使います。インストールなどはLP-techの他の記事を参考にしてみるといいかもしれません。
この記事は畳み込みニューラルネットワークCNNを応用する内容です。
この記事は畳み込みニューラルネットワークCNNを応用する内容です。
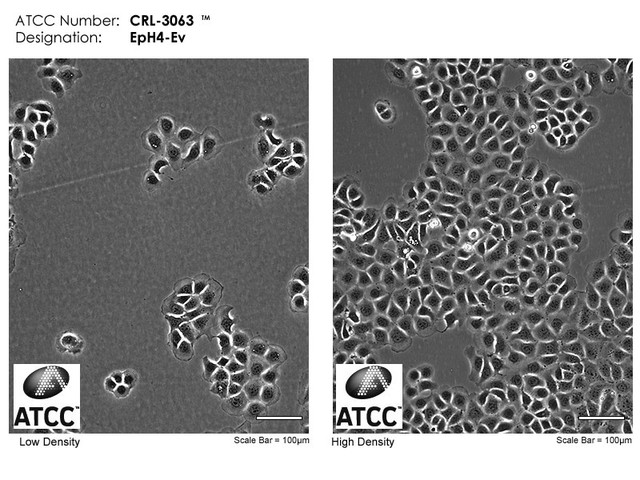
今回は、まず、この2つの細胞の画像のうち右側の高密度の画像をもとに5*5=25枚の分割画像を作ります。次に各25枚すなわち、50枚の画像を3:1で学習用と検証用に分けます。画像数がやや少ないですがデモということで!(途中で画像の数を水増しする処理も紹介しています!!!)
まずはフォルダ構成から。
original_dataには元のデータを入れます。その中の0のフォルダにEpHの画像を、1のフォルダにHEK293の画像を入れています。今回は1枚ずつしかありませんが、何枚入れてもOKです。
画像の分割と明るさの統一
このコードが分割を行う関数になります。
import cv2 import os.path import glob #1枚の画像をN*Nの画像に分割、縮小もする、色補正 def img_split(path,N,label): files = glob.glob(path) files.sort() for i, filename in enumerate(files): #print filename, os.path.isfile(filename) print i, img = cv2.imread(filename, cv2.IMREAD_GRAYSCALE) img = cv2.resize(img,(64*N,64*N)) img = img * 64.0 / cv2.mean(img)[0] height, width = img.shape for x in range(0,N): for y in range(0,N): clp = img[height/N*y:height/N*(y+1), width/N*x:width/N*(x+1)] output = "../train_data/"+label+"/"+str(i*N*N+x*N+y)+'.png' cv2.imwrite(output, clp)
分割と明るさの統一.py
img_split(r"../original_data/0/*.png",5,"0") img_split(r"../original_data/1/*.png",5,"1")
分割.py
画像の水増し
分割しただけでは学習に十分な数の画像とは言えないため、回転によって数を増やします。







 Mochizuki
Mochizuki





