open("C:\\Users\\Hiro\\Downloads\\Rat_Hippocampal_Neuron2.zip");
run("Split Channels");
selectWindow("C3-Rat_Hippocampal_Neuron2.tif");
run("8-bit");
run("Options...", "iterations=1 count=1 black");
setAutoThreshold("Otsu");
//run("Threshold...");
//setThreshold(0, 71);
setOption("BlackBackground", true);
run("Convert to Mask");
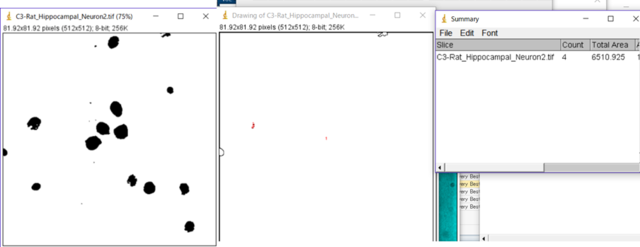
run("Analyze Particles...", " show=Outlines display summarize");パターン①.macro
細胞ではなく背景が輝度値255(=白)で二値化されたためAnalyze Particleがうまく実行されない。(*2)
*2
0が黒、255が白の画像で、Dark Backgroundに✓が入っていないので明るいところがBack(背景)、暗いところが前景として認識されたので、細胞外領域を数えてしまいました。
0が黒、255が白の画像で、Dark Backgroundに✓が入っていないので明るいところがBack(背景)、暗いところが前景として認識されたので、細胞外領域を数えてしまいました。
◆パターン②(Black Backgroundに✓,Dark Backgroundに✓を入れる)
open("C:\\Users\\Hiro\\Downloads\\Rat_Hippocampal_Neuron2.zip");
run("Split Channels");
selectWindow("C3-Rat_Hippocampal_Neuron2.tif");
run("8-bit");
run("Options...", "iterations=1 count=1 black");
run("8-bit");
setAutoThreshold("Otsu");
//run("Threshold...");
setAutoThreshold("Otsu dark");
//setThreshold(90, 255);
run("Convert to Mask");
run("Analyze Particles...", " show=Outlines display summarize");パターン②.macro
細胞が輝度値255(=白)で二値化されたためAnalyze Particleが実行される。(*3)
*3
0が黒、255が白の画像で、Dark Backgroundに✓が入っているので暗いところがBack(背景)、明るいところが前景として認識されたので、細胞が正しく数えられました。
0が黒、255が白の画像で、Dark Backgroundに✓が入っているので暗いところがBack(背景)、明るいところが前景として認識されたので、細胞が正しく数えられました。
◆パターン③(Black Backgroundの✓を外す,Dark Backgroundの✓を外す)
open("C:\\Users\\Hiro\\Downloads\\Rat_Hippocampal_Neuron2.zip");
run("Split Channels");
selectWindow("C3-Rat_Hippocampal_Neuron2.tif");
run("8-bit");
run("Options...", "iterations=1 count=1");
setAutoThreshold("Otsu");
//setThreshold(0, 89);
setOption("BlackBackground", false);
run("Convert to Mask");
run("Analyze Particles...", " show=Outlines display summarize");パターン③.macro
細胞ではなく背景が輝度値255(=黒)で二値化されたためAnalyze Particleがうまく実行されない。(*4)
*4
0が白、255が黒の画像で、Dark Backgroundに✓が入っていないので明るいところがBack(背景)、暗いところが前景として認識されたので、細胞外領域を数えてしまいました。
0が白、255が黒の画像で、Dark Backgroundに✓が入っていないので明るいところがBack(背景)、暗いところが前景として認識されたので、細胞外領域を数えてしまいました。







 エルピクセル編集部
エルピクセル編集部




